Csirke adaptív válasza az alacsony energiájú étrendre: a hipotalamusz lipid anyagcseréjének fő szerepe egy fenotípusos és többszövetű transzkripptikus megközelítéssel
Absztrakt
Háttér
A réteges csirke termelési körülményei változhatnak a hőmérséklet vagy az étrend energiatartalma szempontjából, összehasonlítva az ellenőrzött környezettel, ahol fajtatiszta szelekciót végeznek. Ennek a tanulmánynak az volt a célja, hogy egy többszövetű transzkriptomikus elemzés segítségével jobban megértsük a 15% -os energiahiányos étrend hosszú távú hatásait a tojástermelésre, az energia homeosztázisra és az anyagcserére. A tanulmány célja az volt, hogy összehasonlítsa a táplálkozási beavatkozás hatásait kétrétegű csirkevonalakban, amelyeket eltérően választottak ki a maradék takarmányfelvételre.
Eredmények
Következtetések
Összetett transzkriptóm modulációt figyeltünk meg a csirke hipotalamuszában az alacsony energiájú étrendre reagálva, ami számos változásra utal a szinaptikus plaszticitásban, az endokannabinoid szabályozásában, a neurotranszmisszióban, a lipid anyagcserében, a mitokondriális aktivitásban és a fehérjeszintézisben. Ez a globális transzkriptomikus újraprogramozás megmagyarázhatja az állatok adaptív viselkedési reakcióját (vagyis a takarmánybevitel növekedését) az étrend alacsony energiatartalmára.
Háttér
Eredmények
Az étrend energiaváltása csekély hatással volt a termelési tulajdonságokra, de befolyásolta a takarmányfelvételt és a testösszetételt
Az alacsony energiafogyasztású étrend 14 hetes étrendjét, étrendjét és interakcióit a testsúlyra, a tojástermelésre és a héj erejére, a takarmányfelvételre (FI), a maradék takarmányfelvételre (RFI) és a hasi zsírtömegre az 1. táblázat foglalja össze. az energiatartalom-különbség nem volt hatással a tojástermelésre, azaz a tojásrakás mértékére, a tojás súlyára és a tojás tömegére. Ezzel szemben a testtömeg jelentős csökkenését figyeltük meg a 31. héten (átlagosan mindkét vonalon, - 4,4%, o 1. táblázat: Az eszközök (± SD) és jelentősége a termelés, a takarmány hatékonysága és a testösszetétel tulajdonságai, az étrend, a vonal és kölcsönhatásuk hatása szempontjából
A diéta energiaváltozása transzkripptikus változásokhoz vezet, főleg a hipotalamuszban és a vérben
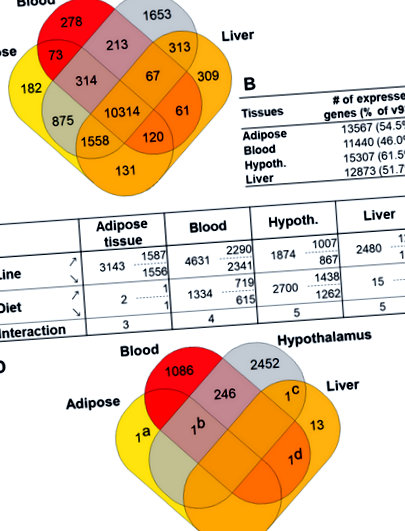
Áttekintés a génexpresszióról és a diéta közötti differenciális expresszióról a zsírszövetben, a vérben, a hipotalamuszban és a májban. a A négy szövetben expresszált és megosztott gének Venn-diagramja. b Az egyes szövetekben expresszált gének teljes száma; zárójelek között, a v87 annotáció százaléka (24 881 gén). c Differenciálisan expresszált gének (DEG) az egyes szövetekben (oszlopok) és az egyes tényezők, vonal, étrend és kölcsönhatás (sorok). Meg kell adni a DEG teljes számát (balra), valamint a fel- (down) és a lefelé expresszált gének (↘) számának részleteit az LE étrendben (vagy R + vonalon) a CT-vel (R-vonalig) összehasonlítva. Hypoth .: Hypothalamus. d A négy szövet étrendje közötti DEG Venn-diagramja. A diagram egyes génjei a következők: a) ENSGALG00000002503 (SFTPA2)
A hipotalamusz transzkriptóm változásainak funkcionális jellemzése az étrend energia kihívásakor
Az étrend energiaváltozására válaszul a hipotalamuszban kimutatott 2700 DEG közül 1438 és 1262 gén volt túl- és alul expresszálódott az LE csoportban a kontrollhoz képest. Ezt a két DEG-listát jellemeztük a KEGG útvonal-kifejezéssel történő dúsítással, a Módszerekben leírtak szerint. A túl- és alul expresszált génlisták esetében 26 és 44 útvonal (oFDR 2. Táblázat: Top 10 (a következő alapján: oFDR) Alul expresszált (A) és a hipotalamuszban túl expresszált DEG (B)
Az alul expresszált génekhez kapcsolódó utak (2A. Táblázat) 91 alul expresszált gént tartalmaztak, amelyek különböző típusú szinapszisokhoz kapcsolódtak: glutamaterg, dopaminerg és GABAerg szinapszisokhoz, valamint a szinaptikus hólyagciklushoz vagy axonvezetéshez. Ezek közül a gének közül különösen GRIA1, GRIA3 és GRIA4 ez kódolja a glutamát receptor alegységeit, az idegrendszerben az uralkodó gerjesztő neurotranszmittert; DDC, - a dopamin szintézisében részt vevő enzim, a jutalmazási rendszerben részt vevő neurotranszmitter kódja, és DRD3 ez kódolja a dopamin receptor alegységét; GABRQ, GABRG2, GABRR2 amely a receptor alegységeit kódolja a gamma-aminovajsavhoz (GABA), a fő gátló neurotranszmitterhez.
A vértranszkriptikus változások funkcionális jellemzése az étrend energiaváltozásakor
Az étrendi változásra válaszul a vérben kimutatott 1334 DEG közül 719, illetve 615 gén volt túl- és alul expresszálódott az LE-ben a CT-csoporthoz képest. A túl- és az alul expresszált DEG-listák KEGG-jellemzése 2, illetve 8 jelentősen dúsított utat tár fel (oFDR 3. táblázat A vérben túl expresszált (A) és alul expresszált DEG (B) -hez társított KEGG útvonalak
A vérben kevésbé expresszált génekhez kapcsolódó útvonalak kapcsolódnak a „Metabolikus utakhoz”, különösen az aminosavak bioszintéziséhez (ACO2, ALDH7A1, CPS1, CTH, ENO2, GOT1, PFKP, TALDO1, TKT, TPI1), a fruktóz és a mannóz anyagcseréje (AKR1B1, AKR1B10, PFKFB4, PFKP, PMM2, TPI1) vagy galaktóz-anyagcsere (AKR1B1, AKR1B10, GALK2, PFKP, PGM2). A koleszterin bioszintézisében részt vevő géneket a vérben alul expresszálták (FDFT1, SQLE, CYP51A1, NSDHL és DHCR24), mint a hipotalamuszban. A túl expresszált génekhez kapcsolódó két út az „RNS lebomlás”, a EDC3, EXOSC5, PABPC1, PAN2, PAN3, PATL1, RQCD1, SKIV2L és TOB2, és a „Ribosoma”, amely 3 RPL-t, 3 MRPL-t, 3 Ribosomális fehérje P oldalsó szár alegységet (RPLP) tartalmazx) és 4 RPS gén, ebből a 13 génből 11 is túl expresszálódott a hipotalamuszban.
Együtt expresszált gének detektálása WGCNA-val a hipotalamusz és a vér DEG listáin belül
A DEG listáinkban található génhalmazok kimutatásához az R csomag WGCNA-t használtuk az együtt expresszált génmodulok azonosításához és klaszterezéséhez (lásd: Módszerek). Amint a 3. ábrán látható, a WGCNA elvált a hipotalamusz (3a. Ábra) és a vér (3c. Ábra) különböző ko-expressziós csoportoktól (színnel jelölt) mind az „LE> CT” (piros színnel), mind az „LE CT” esetében DEG lista és a kék „LE piros és türkiz moduljai” 3. ábra
A hipotalamusz és a vér differenciálisan expresszált génjeire kapott WGCNA modulok elemzése. A hipotalamussal detektált modulok eigengénjeinek hierarchikus csoportosulása (a) és a vér (b) DEG. A modulok színei a modulnevek mellé vannak rajzolva, a modulokban található gének számával. A klaszter nélküli gének a szürke modulban vannak. A jobb oldali négyzetek jelzik, hogy a modul tartalmaz-e túl expresszált (LE> CT) géneket (piros) vagy alul expresszált (LE CT DEG lista). c Az eigengének modulok közötti korrelációs mátrix hőtérképe. Vegye figyelembe a lila és a kék modul (tetejére), valamint türkiz és piros modulok (alsó). d A vér DEG-ből származó két modul-eigengén két terve. Felül: lila és kék modul a LE CT DEG listából. A Δdiet a különbség az LE átlag és a CT átlag között (üres körrel szimbolizálva) az egyes soroknál
Fókuszáljon a differenciálisan expresszált géneket koncentráló genomi régiókra
Három vagy több DEG csoportot kerestünk a fizikai fizikai közelségben (azaz egymás mellett) a genom mentén, amelyeknek szignifikáns páros expressziós korrelációja volt (|r| > 0,7 & oFDR - 4), feltételezve, hogy az ilyen géneket helyi közös mechanizmus szabályozhatja. Két ilyen proximális együtt expresszált géncsoportot találtunk a hipotalamuszban (4a. És b. Ábra), amelyekből összeállt RPS6KA2, MPC1 és SFT2D1 az elsőre (4a. ábra) és C1QA, C1QB és C1QC a másodikhoz (4b. ábra) olyan gének tartoznak, amelyek a fekete WGCNA modulhoz tartoznak, amely immunitással kapcsolatos génekkel gazdagodott.
Az együtt expresszált és a lokalizált gének négy csoportjának genomi lokalizációja és expressziójának páronkénti szóródási sávjai. Minden cselekményben, teteje: a három gén genomi lokalizációja. Alsó: a gének expressziójának (FPKM) páronkénti szétszóródása. a klaszter áll RPS6KA2, MPC1 és SFT2D1. b klaszter áll C1QA, C1QB és C1QC
Vita
Mindkét vonal rétegei alkalmazkodnak az alacsony energiatartalmú étrendhez a takarmányfelvétel növelésével és a test tartalékdinamikájának megváltoztatásával
A petesejt-termelés (szám és tömeg) közötti jelentős különbségek hiánya az LE és a CT csoport között arra utal, hogy az állatok képesek voltak alkalmazkodni a szuboptimális étrendhez. Az állatok által az étrend-energiatartalom csökkenésének kompenzálására alkalmazott adaptív mechanizmusok a takarmánybevitel növekedését és a hasi zsírszövet csökkenését jelentették. A takarmányfelvétel növekedése a 15% -os energiahiányos étrendre adott válaszként 14 hét alatt összhangban áll Grobas és mtsai. [1] és Harms és mtsai. [2]. Ez a megnövekedett fogyasztás azonban nem tette lehetővé, hogy az LE csoportból származó rétegek teljes mértékben ellensúlyozzák az energia különbségeket (1. táblázat), amint azt az étrendcsoportok közötti energiafogyasztás jelentős különbsége jelzi. A zsírtömeg százalékos arányának az össztömegre való csökkenése, valószínűleg ennek a hiányos kompenzációnak köszönhető, összhangban áll a Murugesan és Persia által közölt eredményekkel [3], ahol a rétegeket 3% -os energiahiányos étrenddel etették a kontrollhoz képest. 11 hét alatt, bár a szerzők nem figyeltek meg takarmánybeviteli változást, talán a két étrend közötti kis energiakülönbség miatt.
Az expressziós szinten a vonal × diéta közötti jelentős interakció hiánya összhangban áll a tulajdonság szintjén történő interakció hiányával, vagyis mind az R +, mind az R-madarak hasonló módon és ugyanolyan mértékben reagálnak az energiahiányos étrendre. Az expresszió szintjén a 3e. Ábra Δdiet értékei szemléltetik ezt a következtetést: Például a lila modulhoz tartozó gének Δdietje hasonló a két vonalon, míg ezek a gének inkább R-, mint R+.
Az alacsony energiatartalmú étrend nem befolyásolta a máj és a zsírszövet transzkripcióit
A vérsejtek részt vesznek a CT és az LE diéta változásaihoz való alkalmazkodásban
A hipotalamuszban az alacsony energiatartalmú étrend úgy tűnik, hogy megváltoztatja az általános szinaptikus szervezetet, részben a koleszterin modulációja és a zsírsavakhoz kapcsolódó globális fehérjeszintézis révén. β-oxidáció
A hipotalamusi arachidonsav részt vehet a takarmányfelvétel különbségében az LE és a CT csoportok között a hipotalamusz endokannabinoid és komplement rendszereket magában foglaló mechanizmusokon keresztül.
Javasolt mechanizmus, amely megnöveli a takarmányfelvételt az LE étrendben. A diétás zsírsavakat az FADS1, FADS2, ELOVL5 és FADS1, arachidonsav (AA) termeléséhez vezet. Az arachidonsav végül a NAPE-PLD hatásának köszönhetően arachidonoyil-etanol-amin (AEA) termeléséhez vezet. Az AEA hat a CB1R-re, ami a takarmánybevitel növekedéséhez vezet. FADS1 és 2: Zsírsav-deszaturáz 1 és 2, ELOVL5: Nagyon hosszú láncú zsírsavak 5-ös fehérje megnyúlása, NAPE-PLD: N-acil-foszfatidil-etanol-amin-foszfolipáz D, CB1R: 1. kannabinoid-receptor, AA: arachidonsav, PE: foszfatidil: N-arachidonoil-foszfatidil-etanol-amin, AEA: Arachidonoyil-etanol-amin (más néven: Anandamid)
Következtetések
Mód
Állatok és étrend
Szövetmintavétel
A 31. héten az egyes vonalakból (R- és R +) és az egyes étrendekből (CT és LE) nyolc állatot választottak ki a levágásra szolgáló csoport reprezentatív képviselőjeként, azaz 8 × 2 × 2 = 32 állat. A rétegeket etetett állapotban vágták le nyakvágással és vérzéssel, közvetlenül a fej elektromos kábítása után. Közvetlenül a levágás után a hasi zsírszövetből, a bal májlebeny végéből és a hipotalamuszból mintát vettünk, folyékony nitrogénben lefagyasztottuk és -80 ° C-on tároltuk az elemzésig. Ugyanezen állatok vérmintáit gyűjtöttük az occipitalis sinusból EDTA csövekbe, és 100 μL vért eltávolítottunk, és 1 ml TRIzol® reagenssel hígítottunk (Invitrogen, Kalifornia, USA). Erőteljes keverés után a csövet öt percig szobahőmérsékleten tartjuk, majd folyékony nitrogénben gyorsan lefagyasztjuk és -80 ° C-on tároljuk az RNS-ek kivonásáig.
Jellemzők gyűjtése és elemzése
A 45 R + (34 CT és 11 LE) és 51 R- (36 CT és 15 LE) madarak esetében a teljesítményhez és a testösszetételhez kapcsolódó hét tulajdonságot rögzítettünk. A tojásszámot az első tojás (kb. 21 hetes életkor) dátumától a 31. hét koráig és a tojásrakás mértékét (azaz a felvételi időszak alatt rakott tojások számát elosztva a napi időszak hosszával,% -ban kifejezve) rögzítettük. kiszámították; a tojás súlyát (g), a statikus merevséget (N.mm - 1) 30 tojásonként gyűjtött tyúkról 3 tojásból számoltuk, és a hasi zsírokat levágáskor súlyoztuk. A heti takarmányfelvételt 4 héten keresztül mértük, 27-től 31-ig és 31 hetes testtömegig (g). A maradék takarmányfelvételt Bordas et al. [4]. A tulajdonságokat R 3.4.2 verzióval elemeztük [46]. Kétirányú varianciaanalízist hajtottak végre a vonallal, az étrenddel, valamint a vonal és az étrend közötti kölcsönhatással, mint fő hatásokkal az lm R funkcióval és az R csomag „autóval” [47].
RNS izolálás
Körülbelül 100 mg zsírszövetet és 30 mg májat homogenizáltunk TRIzol® reagensben (Invitrogen, Kalifornia, USA), és az 1 ml TRIzol®-val kevert teljes vért 4 és 4,5 között állítottuk be 10 μl 5 N jégecettel. sav [48]. Ezután a teljes RNS-t a gyártó utasításainak megfelelően extraháltuk, 50 μl RNS-mentes vízben újraszuszpendáltuk és -80 ° C-on tároltuk. A hipotalamuszhoz az Allprep DNS/RNS (Qiagen) kitet használtuk. Az RNS-t a gyártó utasításai szerint kivonták a hipotalamuszból. A teljes RNS-t NanoDrop® ND-1000 spektrofotométerrel (Thermo Scientific, Illkirch, Franciaország) számszerűsítettük. Az RNS minőségét egy Agilent 2100 bioanalizátorral (Agilent Technologies France, Massy, Franciaország) ellenőriztük. Az átlagos RNS-integritási számok a zsírszövet esetében 7,3 ± 0,6 (átlag ± SD), a hipotalamusznál 8,8 ± 0,48, a teljes vér esetében 8,2 ± 0,5 és a máj esetében 9,2 ± 0,3.
RNS-seq adatgyűjtés
A páros végű szekvenálást minden mintán Illumina HiSeq3000 (Illumina, Kalifornia, USA) rendszerrel végeztük, 2 × 150 bázispárral. Az átlagosan 465 bp méretű inszertet tartalmazó könyvtárakat az Illumina utasításait követve készítettük el a poli-A RNS-ek tisztításával (TruSeq RNS Sample Prep Kit). Az indexelő címkéket tartalmazó illumina adaptereket adtuk hozzá a minták későbbi azonosításához. A mintákat PCR-amplifikáltuk, majd kvantitatív PCR-t hajtottunk végre a könyvtár kvantifikálásához (QPCR NGS Library Quantification kit). Nyolc mintát töltöttünk egy sávon egy áramlási cellában, 2 mintával a négy vonal × diéta csoport mindegyikéhez, hogy minimalizáljuk a sávok közötti torzítást. Szekvenálás után az indexelt adapter szekvenciákat a CASAVA v.1.8.2 szoftverrel (Illumina) vágtuk le. Mintánként átlagosan 90 millió leolvasást kaptunk (84 millió a zsírszövetre, 98 millió a vérre, 86 millió a hipotalamuszra és 90 millió a májra), összesen 11 milliárd olvasás. Minden minta esetében az olvasmányokat hozzárendeltük a Gallus gallus-5 referencia genom a STAR v.2.3.0e alkalmazásával [49]. A PCR duplikátumokat eltávolítottuk a SAMtools csomag rmdup eszközével [50]. Minden egyes mintához a mennyiségi meghatározást RSEM [51] alkalmazásával hajtottuk végre, Ensembl v93 annotációval.
RNS-seq adatok elemzése
Az összes elemzést az 3.4.2 R verzióval hajtottuk végre. Az M-értékek (TMM) skálázási tényező metszett átlagát használtuk a könyvtár méretének normalizálására [52] az R/Bioconductor csomag edgeR [53] 3.12.1 változatának felhasználásával. Mindegyik szövetben kiválasztottuk az expresszált géneket, ha FPKM-expressziójuk 0,1-nél nagyobb volt a csoportvonal × étrend mintáinak legalább 80% -ában (az FPKM-expressziót TMM normalizálás után kaptuk, a edgeR csomagtól kapott „rpkm” függvény segítségével). A differenciál expressziós elemzést az R/Bioconductor csomag edgeR [53] felhasználásával hajtottuk végre, egy általánosított negatív binomiális modell alapján, a modell illesztéséhez. A géndiszperziós paraméterek becslésénél a „edgeR-Robust” módszert alkalmaztuk a potenciális kiugró értékek figyelembevételéhez [54]. P-az értékeket többszörös teszteléssel korrigáltuk a Benjamini-Hochberg-féle megközelítéssel [55] a hamis felfedezési arány (FDR) ellenőrzésére, és azonosítottuk, hogy a gének szignifikánsan eltérő módon expresszálódtak-e, ha oFDR 0,7 és oFDR - 4).
RT-qPCR elemzés
Az adatok és anyagok rendelkezésre állása
A 64 RNS-seq minta elérhető az Európai Nukleotid Archívumban (ENA) az ENA sorozat PRJEB28745 csatlakozási számán keresztül.
- A magas fehérjetartalmú teljes étrend-pótlás energia-anyagcserére, anyagcserére gyakorolt hatásainak vizsgálata
- A diéta fontos szerepet játszik az anti-PD-1 rák immunterápiás MDedge válaszában
- Fogyókúra és megelőző szerepe a prosztata betegségben - Kivonat - European Urology 1999, Vol
- Fogyókúra összehasonlítása WW és a gyors anyagcsere diéta; Joely Sue Burkhart
- Energia étrend - Eco Slim